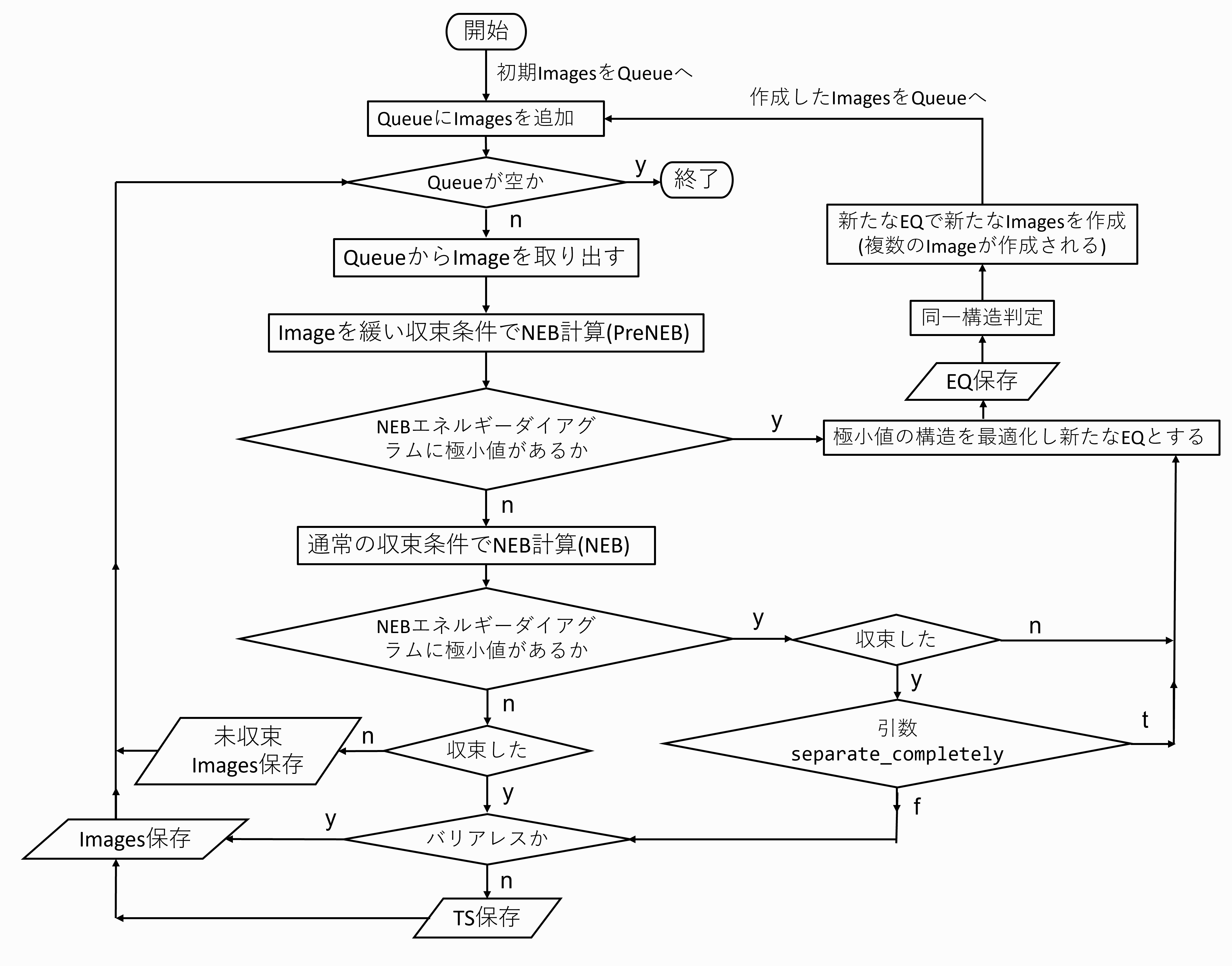
PinNEB計算
PinNEBの概要
PinNEBはNEBエネルギーダイアグラム中に谷間を見つけたら最適化し,素反応に切り分けて計算を行なう.
次のステップで行う
STEP1 : 通常のNEB計算を行なう
STEP2 : STEP1のエネルギーダイアグラム中に谷間(極小点)があれば最適化し, 新たなEQとする
STEP3 : STEP2で見つけたEQ(中間体)を経由する新たなNEBイメージを作成し,STEP1,STEP2を行なう
STEP4 : 極小点がなくなるまで(つまり素反応が見つかるまで)STEP1,2,3を繰り返す
より詳細なフローはこのページの最後に示した.
基本操作
from grrmpy.automate import Queue,PinNEB
pin_neb = PinNEB(images,constraints=[])
pin_neb.run()
細かいパラメータを指定する
例えば極小点があってもNEBが収束すれば,NEBイメージを分解しないようにする場合, separate_completely をFalseにする.
>>> pin_neb.set_param(separate_completely=False)
その他指定可能な引数は
PinNEB を参照する.また,指定可能な全ての引数は
get_param() で確認できる.>>> pin_neb.get_param()
PinNEBは
Queue クラスを用いてQueueの管理を行なっている.Queueはデフォルトでは幅優先でQueueの取り出しを行なう(priority=0)が, PinNEBの場合priority=1に設定すると,
反応熱の高い順にQueueからイメージが取り出される
>>> pin_neb = PinNEB(images,constraints=[],priority=1)
同一構造判定
PinNEBでは不必要なNEB計算を防ぐために構造最適化後の構造について同一構造判定を行なっている.
例えばEQ0とEQ1をINI,FIN構造としNEB計算をした際に1つの極小値が見つかり,この最適化後の構造をEQ2とする.
通常であれば,EQ0とEQ2, EQ2とEQ1で新たにNEB計算をすれば良いが,もしEQ0とEQ2が同一構造であった場合,NEB計算する意味はない.
それどころか,同一構造でNEB計算をするとNEBエネルギーダイアグラムが歪な形状になり,バリアレス判定を行ないにくくなってしまう.
PinNEBでは
is_similar で同一構造判定を行なっている が
自作の同一構造判定関数を設定することもできる.atoms1,atoms2の引数をとり,同一構造の場合True,異なる場合Falseを返す関数を定義する.
例えば,2つの構造のエネルギー差が3 kJ/mol以下の場合同一構造とする場合
from ase.units import mol,kJ
def user_function(atoms1,atoms2,energy=3):
e1 = atoms1.get_potential_energy()
e2 = atoms2.get_potential_energy()
if (abs(e1-e2)*mol/kJ) <= energy:
return True
else:
return False
このような関数を定義し,
>>> pin_neb.set_param(isomorphism_func=user_function)
set_param で適用する事ができる.
Path.datについて
計算中にPath.datが更新され,これに基づいてPath.html(反応座標)が作成される
Path.datの中身の例を示す
[Dir]
EQ: ./EQ_List/
TS: ./TS_List/
[path]
path: EQ0|EQ3;EQ4~EQ2;TS0;EQ1
このPath.datから次のグラフが作成される.
EQ_List,TS_Listに保存されているtrajファイルを読み込みグラフを作成が作成される.
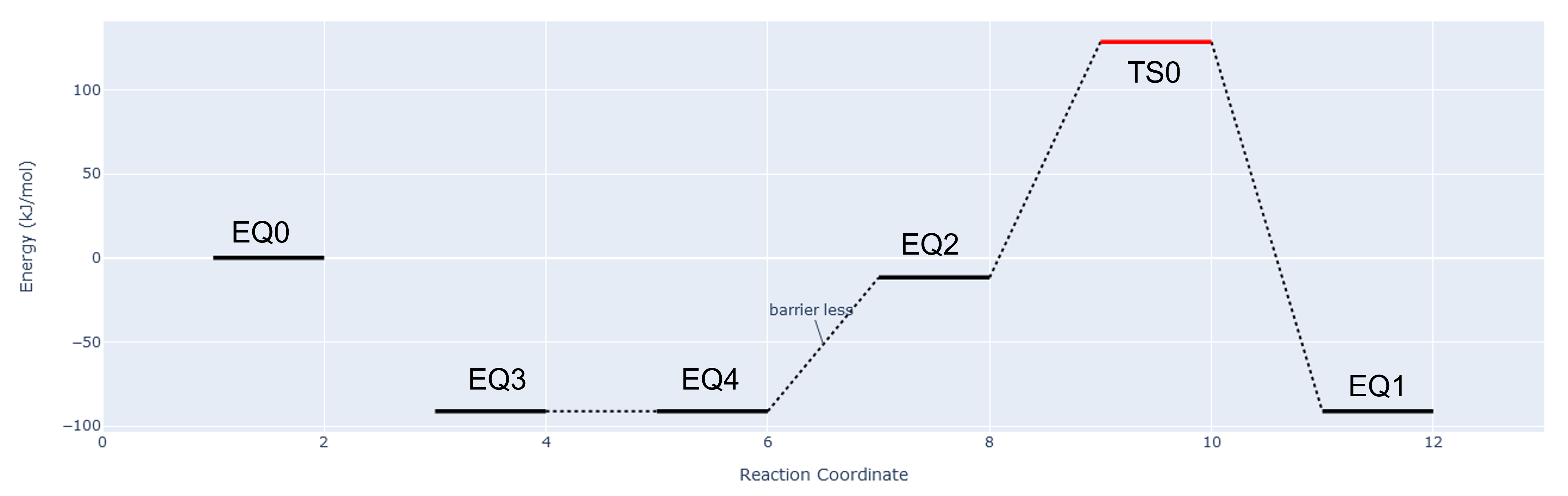
記号 |
意味 |
|---|---|
; (セミコロン) |
破線でつなぐ(パスがつながっている) |
| (バーティカルバー) |
線を書かない(パスがつながていない) |
~ (チルダ) |
バリアレス 破線でつなぐ&barrier lessと表示 |
Path.datに~が一つしか書かれていないのにhtml上に"barrier less"のラベルが複数表示されるバグが存在する.
これはPlotly側の既知のバグから生じている.
notebook上で
>>> from grrmpy.path import create_reactpath
>>> rpath = create_reactpath("Path.dat")
>>> rpath.write_html("Path.html")
とすれば,正しいグラフに書き直すことができる.
PinNEBのフロー