Automateモジュール
PinNEB
- class grrmpy.automate.pin_neb.PinNEB(images, constraints=[], parallel=True, mic=None, calc_func=<function pfp_calculator>, eq_folder='EQ_List', logfolder='log', images_folder='Images_List', unconv_folder='unconv', ts_folder='TS_List', errorfile='ERROR', path_html='Path.html', path_dat='Path.dat', priority=0, maxjob=20)[ソース]
- get_param()[ソース]
Parameters:
- separate_completely: bool
- 通常のNEBで収束した時,エネルギーダイアグラム上に極小点があれば,極小点を最適化してNEB分割する.
- minenergy: float
- minenergy(kJ/mol)以下の活性化エネルギーの場合は極小点があっても無視する.
- barrier_less: float
- barrier_less(kJ/mol)以下の活性化エネルギーの場合,バリアレスとみなす.
- nimages: int or tuple
- intので指定した場合,指定した数のイメージ数で新たなNEBイメージを作成する.tupleで指定する場合,3要素(a,b,c)のタプルを与える.aÅ毎にイメージを作成する.最大でもb個,最低でもc個のイメージを作成する.
- isomorphism_func: function object
- 同一構造判定を行なう関数,デフォルトではis_similarを用いる.
- rename_preneb_file: bool
- Trueの場合,pre_NEBファイルは消す
- preneb_optimizer: Optimizer
PreNEBのOptimizer
- preneb_climb_list: list of bool
- climbのリスト
- preneb_maxstep_list: list of float
- maxstepのリスト
- preneb_automaxstep_climb_false:
- {1:0.05, 0.5:0.1, 0.1:0.2, 0.05:0.3, 0:0.35}
- preneb_automaxstep_climb_true: dict
{0.1:0.03,0:0.01}
- preneb_fmax_list :list of float
- preneb_steps_list: list of int
- preneb_kwargs:
- prenebopt_kwargs:
- neb_optimizer: Optimizer
- "neb_climb_list": list of bool
- neb_maxstep_list:
- [0.02,0.05,0.1,None,0.01,None],
- neb_automaxstep_climb_false:
- {1:0.05, 0.5:0.1, 0.1:0.2, 0.05:0.3, 0:0.35},
- neb_automaxstep_climb_true":
- {0.1:0.03,0:0.01},
- neb_fmax_list:
- [0.01,0.05,0.05,0.05,0.05,0.05],
- neb_steps_list:
- [100,200,200,2000,500,1500],
- neb_kwargs:
- {}
- nebopt_kwargs:
- {}
- opt_optimizer:
- DEFAULT_OPTIMIZER
- opt_maxstep_list:
- [0.02,0.05,0.1,0.2]
- opt_automaxstep:
- {1:0.1, 0.5:0.2, 0.1:0.3, 0.05:0.4, 0:0.5}
- opt_fmax_list:
- [0.001,0.001,0.001,0.001]
- opt_steps_list:
- [50,100,300,10000]
- opt_kwargs:
- {}
- same_energy_and_fmax_arg:
- {"times":10,"row":2,"force_digit":4,"energy_digit":6},
- steep_peak_arg:
- {"max_ea":800, "nsteps":100}
AutoPinNEB
- class grrmpy.automate.pin_neb.AutoPinNEB(eq_list, connections, constraints=[], parallel=True, mic=None, priority=0, maxjob=20, calc_func=<function pfp_calculator>, eq_folder='EQ_List', ts_folder='TS_List', images_folder='Images', unconv_folder='unconv', logfolder='log', error_file='ERROR', path_file='AllPath.dat')[ソース]
- get_param()[ソース]
Parameters:
- separate_completely: bool
- 通常のNEBで収束した時,エネルギーダイアグラム上に極小点があれば,極小点を最適化してNEB分割する.
- minenergy: float
- minenergy(kJ/mol)以下の活性化エネルギーの場合は極小点があっても無視する.
- barrier_less: float
- barrier_less(kJ/mol)以下の活性化エネルギーの場合,バリアレスとみなす.
- nimages: int or tuple
- intので指定した場合,指定した数のイメージ数で新たなNEBイメージを作成する.tupleで指定する場合,3要素(a,b,c)のタプルを与える.aÅ毎にイメージを作成する.最大でもb個,最低でもc個のイメージを作成する.
- isomorphism_func: function object
- 同一構造判定を行なう関数,デフォルトではis_similarを用いる.
- rename_preneb_file: bool
- Trueの場合,pre_NEBファイルは消す
- preneb_optimizer: Optimizer
PreNEBのOptimizer
- preneb_climb_list: list of bool
- climbのリスト
- preneb_maxstep_list: list of float
- maxstepのリスト
- preneb_automaxstep_climb_false:
- {1:0.05, 0.5:0.1, 0.1:0.2, 0.05:0.3, 0:0.35}
- preneb_automaxstep_climb_true: dict
{0.1:0.03,0:0.01}
- preneb_fmax_list :list of float
- preneb_steps_list: list of int
- preneb_kwargs:
- prenebopt_kwargs:
- neb_optimizer: Optimizer
- "neb_climb_list": list of bool
- neb_maxstep_list:
- [0.02,0.05,0.1,None,0.01,None],
- neb_automaxstep_climb_false:
- {1:0.05, 0.5:0.1, 0.1:0.2, 0.05:0.3, 0:0.35},
- neb_automaxstep_climb_true":
- {0.1:0.03,0:0.01},
- neb_fmax_list:
- [0.01,0.05,0.05,0.05,0.05,0.05],
- neb_steps_list:
- [100,200,200,2000,500,1500],
- neb_kwargs:
- {}
- nebopt_kwargs:
- {}
- opt_optimizer:
- DEFAULT_OPTIMIZER
- opt_maxstep_list:
- [0.02,0.05,0.1,0.2]
- opt_automaxstep:
- {1:0.1, 0.5:0.2, 0.1:0.3, 0.05:0.4, 0:0.5}
- opt_fmax_list:
- [0.001,0.001,0.001,0.001]
- opt_steps_list:
- [50,100,300,10000]
- opt_kwargs:
- {}
- same_energy_and_fmax_arg:
- {"times":10,"row":2,"force_digit":4,"energy_digit":6},
- steep_peak_arg:
- {"max_ea":800, "nsteps":100}
is_similar
- grrmpy.automate.pin_neb.is_similar(atoms1, atoms2, energy_dif=10, bond_dif=(1, 1.0), mult=1, **kwargs)[ソース]
atoms1とatoms2を同一構造とみなすか(NEBでつなぐ必要はないか?)
PinNEBの引数:isomorphism_funcのための関数
基本的には結合状態が同じ且つ,エネルギー差がenergy_dif(kJ/mol)以下であれば同一構造とみなす. また,bond_dif[0] 本以下の結合状態の違いであり,その結合の差がbond_dif[1] Å以下の時も同一構造とする.
Parameters:
- atoms1: Atoms
Atomsオブジェクト
- atoms2: Atoms:
Atomsオブジェクト
- energy_dif: float
同じ結合状態かつ,エネルギー差がenergy_dif(kJ/mol)の時,同一構造とする
- bond_dif: Tuple[int,flaot]
- 1要素目は許容できる結合の差(結合本数)2要素目は結合状態の異なる部分の許容できる結合距離の差(Å)
SinglePath
SinglePathはSNEB( grrmpy.neb.auto_neb.SNEB ),振動数計算,IRC計算をiniとfinが繋がるまで繰り返す.
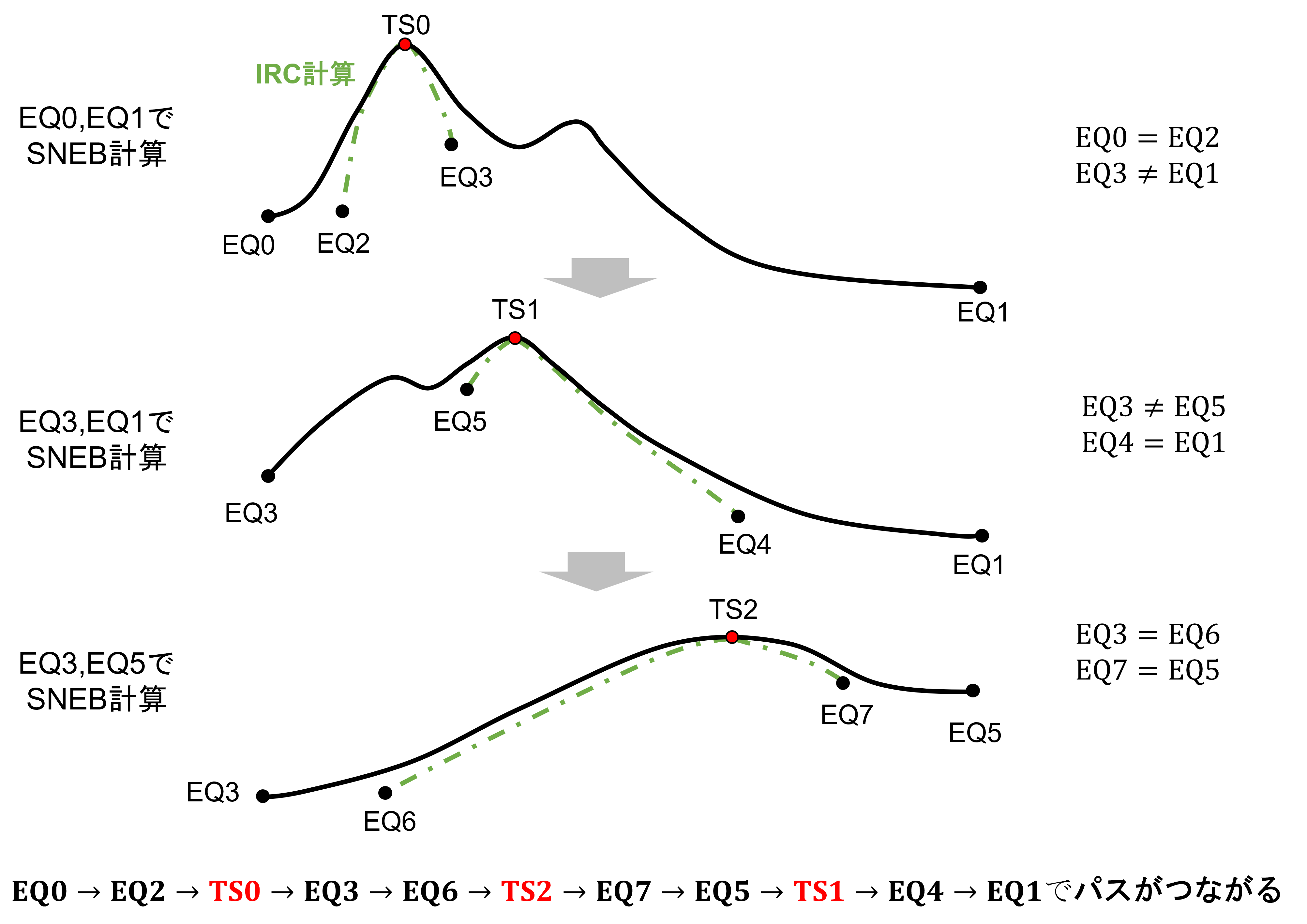
- class grrmpy.automate.by_neb.SinglePath(*data, indices=None, parallel: bool = True, mic: ~typing.Optional[bool] = None, calc_func=<function pfp_calculator>, debug=False, constraints=[])[ソース]
Parameters:
- *data:
- 3種類の方法でinput構造を与えることができる.
- ini,fin構造と初めの中間イメージの数を与える(3つの引数を与える)
>>> sp = SinglePath(ini,fin,21,indices=[1,2,3,4]) # 21個内部にイメージを作成
- ini,fin構造を与える(2つの引数を与える.初期イメージの数は13になる)
>>> sp = SinglePath(ini,fin,indices=[1,2,3,4])
- images構造を与える(1つの引数を与える.calculatorを付ける必要はない)
>>> sp = SinglePath(images,indices=[1,2,3,4])
Constraintsがある場合
>>> f = FixAtoms(indices=[0,1,2,3]) >>> sp = SinglePath(ini,fin,indices=[1,2,3,4],constraints=f)
>>> f = FixAtoms(indices=[0,1,2,3]) >>> c = FixBondLength(0, 1) >>> sp = SinglePath(ini,fin,indices=[1,2,3,4],constraints=[f,c])
imageの数は奇数個を推奨する.
- indices: list
振動数計算を行なう時に動かす原子のindex番号のリスト
- parallel: bool
並列計算を行なう場合True.
- mic: bool
- 最小イメージ規則を適用する場合True.Noneの場合,周期境界条件で計算する場合自動でTrueにする.デフォルトはTrue.
- calc_func: function object
calculatorを返す関数. デフォルトは
pfp_calculator- debug: bool
debug.logを出力する(デバック用)
注釈
EQ_list.traj, TS_list.trajが既にディレクトリ中にある場合,上書きされてしまうので1つフォルダ内で複数の計算を行なわないようにする!!!- property default_param
run()のデフォルトのパラメータを返す.
General:
- stopping_criterion: int
停止基準. SNEBの計算をstopping_criterion回行なうと停止する.
- struct_check_threshold: list of float
- 2つの構造が同一構造であるとみなす基準.a,b,c,d,e,fの6つの要素を指定する.基準1,基準2のどちらかを満たしている時,同一構造とする.基準1:電子エネルギーの差, 原子間距離のRMS誤差,原子間距離の最大距離がそれぞれa kJ/mol, b%, cÅ 以下の時, 同一構造であるとみなす基準2:エネルギーの差がd kJ/mol以下であり,indicesで指定した原子を重ね合わせた時,(最小二乗法で重ねる)原子間距離のRMS誤差,原子間距離の最大距離がそれぞれ,e%, fÅ 以下の時, 同一構造であるとみなす.
- minimize_rotation_and_translation: boolean
- 基準1で2つの構造の座標が近くなるように再配置して解析する場合Ture.(最小二乗値が小さくなるよう再配置を行なう)
SNEB:
- optimizer: Optimizer class
NEB計算に使用するOptimizerの種類
- nimages: list of int
各NEB計算の中間イメージの数
- maxstep: list of float
- 各NEB計算でのmaxstep値のリストNoneの場合,forceに合わせて自動的にmaxstepsを変化させる.
- fmax: list of float
各NEB計算でのfmax値のリスト
- steps: list of int
各NEB計算でのsteps値のリスト
- tolerance: float
NEBバンド中のtolerance(kJ/mol)以下の谷間はEQとは考えないで計算する.
- threshold: list of float
活性化エネルギーに対してthreshold%以下の点は削り新たなNEBイメージを作成して計算する
- dist: float
- 新たなNEBイメージを作成する際,TSの両隣に,TSからdistÅ離れた位置にイメージを作成する.TS付近にイメージを密集させるために行なう.
- min_nimages: int
- 新たなイメージを作成するにあたって,それまでのNEBを削ることになるが,少なくともmin_nimages個は確保するようにする.
- with_stop: bool
- optimizerにstop機能を付ける場合True.max_n回連続でEnergyとForceが同じ状況がtimes回あった時計算を終了する.grrmpy.optimize.functions.add_stop_to()を参照
- max_n: int:
with_stop=Trueの場合に指定. with_stop(add_stop_to)の引数. デフォルト12
- times: int
with_stop=Trueの場合に指定. with_stop(add_stop_to)の引数. デフォルト2
- climb_steps: integer
- climb=Falseで収束しなかった場合,その後のTrueでのNEB計算のStep数をclimb_steps回にする.Noneの場合,climb=Falseの時と同じstepsで計算する.一番最後のNEB計算ではclimb_stepsに関係なくclimb=Falseの時と同じstepsで計算する.
- climb: bool
- Falseの場合,climbはFalse,False,False,False...Trueで行なう.Trueの場合はclimbはFalse,True,Fase,True,False....Trueで行なうFalseにした場合,climb_stepsを設定する意味はなくなる.
IRC:
- optimizer1: Optimizer calss
振動数計算後のエネルギーダイアグラム解析の結果,TSがはっきり分かる場合に使用するOptimizer
- optimizer2: Optimizer calss
振動数計算後のエネルギーダイアグラム解析の結果,TSがはっきり見えない場合に使用するOptimizer
- maxsteps:float or None
- maxsteps値. maxstepsの値を大きく設定したとしても,始めの200回はmaxstep=0.03で計算する.Noneを指定した場合,forceに合わせて自動的にmaxstepを調整する.
- dif: float
- 振動数計算後のエネルギーダイアグラム解析の結果,TS(極大値)と谷間(極小値)の差が,dif(kJ/mol)以下の時,optimizer2を使って計算する.
- calc_notop_ts: boolean
- 振動数計算後のエネルギーダイアグラム解析の結果,TSがない(極大値がない)時,IRC計算を行なわないならFalse.
AutoOpt
基本的な使用方法
from grrmpy.io import log2atoms
from grrmpy.automate import AutoOpt
from grrmpy.optimize import FIRELBFGS
eq_list = log2atoms("XXX_EQ_list.log","XXX.com","POSCAR")
opt = AutoOpt(eq_list,optimizer=FIRELBFGS)
opt.run()
計算後のデータの取り出し方
計算後にはStructuresフォルダに最適化後の構造が保存されるので
from grrmpy.io import read_traj
opted_eq_list = read_traj("Structures")
で構造を取り出せる.
細かな条件を設定する方法
細かい計算条件は run の引数で設定する. (maxstep=0.02,steps=200)→(maxstep=0.05,steps=500)→(maxstep=0.2,steps=1000)の条件で計算する場合.
opt.run(maxstep_list=[0.02,0.05,0.2],steps_list=[200,500,10000])
maxstep_list と steps_list の要素数が一致していないとエラーになるので注意する
続きから計算する場合
opt.run(restart=17) # EQ17から計算を開始する
- class grrmpy.automate.auto_opt.AutoOpt(atomslist, optimizer=<class 'ase.optimize.lbfgs.LBFGS'>, constraints=[], trajectory=False, logfile=True, calc_func=<function pfp_calculator>, errorfile='ERROR', traj_foldername='trajectory', log_foldername='log', save_foldername='Structures')[ソース]
最適化後の構造は'Structure'フォルダ内にtrajファイルで保存される.
計算後の構造を一括で読み込むには
>>> import grrmpy.io import read_traj >>> atoms_list = read_traj('Structure')
Parameters:
- atomslist: list of Atoms
Atomsのリスト
- optimizer: object
使用するOptimizer.デフォルトはLBFGS.
- constraints: ASE constraint
- FixAtoms等の制約.複数設定する場合はリストで与える.eq_list中のAtomsに既にconstraintがある場合,改めて設定する必要はない.
- trajectory: bool
- Trueの場合,最適化途中の構造をtrajに保存する.'trajectory'フォルダー内に保存される.
- logfile: bool
- Trueの場合, logファイルを保存する.'log'フォルダー内に保存される.
- calc_func: object
calculatorを返す関数
- run(maxstep_list=[0.05, 0.2], steps_list=[200, 10000], fmax=0.001, indexes=None, restart=None, **kwargs)[ソース]
Parameters:
- maxstep_list: float or list of float
- maxstep.optimizeをFIRELBFGSにした場合,maxstep_fire,maxstep_lbfgsどちらもmaxstepで指定した値になる.
- steps_list: int or list of int
steps
- fmax: float
収束条件
- indexes: list of int
- index番号で指定した構造のみを計算するindexesとrestartは同時に指定できない.
- restart: int
- 指定した番号から計算を開始する.indexesとrestartは同時に指定できない.
- kwargs:
その他,optimizer(LBFGSなど)ののインスタンス引数を指定できる.
AutoNEB
- class grrmpy.automate.auto_neb.AutoNEB(eq_list, connections, constraints=[], parallel=True, mic=None, calc_func=<function pfp_calculator>, savefolder='Structures', unconvergedfolder='unconv', logfolder='log', htmlfolder='html', errorfile='ERROR')[ソース]
連続的にNEB計算を行なう
attach(),attach_stop(),update(),next_i()に関数を与える事ができる.attach()とattach_stop()はイタレーション毎に実行される.update()はNEB計算が終了する毎に実行される.next_i()は1サンプルの計算を終える毎に実行される.STOP.txtファイルをディレクトリ中に設置すると計算を一時停止,またはスキップできる.STOP.txtの中にSTOP = TUREとすると,計算が一時停止される.SKIP = TRUEとすると現在実行中サンプルの計算が中止される.STOP.txtで停止を行なうとRESTART.txtが作成される.ディレクトリ中にRESTART.txtが存在する場合,途中から再開する.Parameters:
- eq_list: list of Atoms
- EQのリスト(Atoms)
- connections:
- CONNECTiONS情報Ex) [[0,1],[2,4],[3,5],...]
- constraints: Constraintオブジェクト
- FixAtomsなどの制約.
- parallel: bool
- NEBで並列計算を行なう場合True.
- mic: bool
- 最小イメージ規則を適用する場合,True.デフォルトでは周期境界条件で計算する場合自動でTrueにする.
- calc_func: function object
- calculatorを返す関数
- savefolder: str
- 収束した構造を保存するためのフォルダ
- unconvergedfolder: str
- 収束しなかった構造を保存するためのフォルダ
- logfolder: str
- logファイルを格納するフォルダ
- htmlfolder:
- エネルギーダイアグラムのhtmlを保存するためのフォルダ
- errorfile:
- ERRORファイルのパス計算中にエラーが発生した場合,ERRORファイルに書き込まれる.
- attach_stop(*args, **kwargs)[ソース]
- grrmpy.optimize.optimizer.OptWithCondのattach_stopと同じTrue,Falseを返す関数を設定する.イタレーション毎に関数が実行されTrueを返した時に計算が停止する
- property default_param
run()の際に使用されるデフォルトの引数.
climb_list,maxstep_list,fmax_list,steps_listの要素数は一致している必要がある.
- base_optimizer: Optimizer
- NEB計算に使用するOptimizer.ここで指定したOptimizerを継承して作成されるOptWithCondオプティマイザーが実際には使用される.
- climb_list: list of bool
毎回のNEB計算のclib
- maxstep_list: list of float
- 毎回のNEB計算のmaxstep_list要素にNoneを指定した場合,forceに合わせて自動でmaxstepを変化させる.どのように変化させるかはautomate_maxstep_true,automate_maxstep_falseで指定できる.
- fmax_list: list of float
- 毎回のNEB計算のfmax
- steps_list: list of int
- 毎回のNEB計算のsteps
- neb_kwargs: dict
- NEBのインスタンス時の引数を指定したい場合に用いる例えばばね定数kを変えたい場合{"k"=0.2}のようにするここで指定した値は全てのNEB計算に適用される
- opt_kwargs: dict
- Optimizerのインスタンス引数を指定したい場合に用いる.
- automate_maxstep_true: dict
- climb=True時にautomaxstepを使用した際のmaxstep値
- automate_maxstep_false: dict
- climb=False時にautomaxstepを使用した際のmaxstep値
- same_energy_and_fmax_arg: dict
- 停止基準
- steep_peak_arg:
- 停止基準
- run(nimages, restart=0, **param)[ソース]
Parameters:
- nimages: int or list of int
NEBのイメージの数. listで指定した場合はサンプル毎に違うイメージ数でNEB計算できる.
その他の引数はdefault_paramで確認する
サンプル
>>> auto_neb.run(16,steps_list=[100,1000,1500])
以下のコードでも同じ処理を行なえる
>>> param = auto_neb.default_param >>> param["steps_list"] = [100,1000,1500] >>> auto_neb.run(16,**param)
途中から計算を再開するには
>>> auto_neb.run(16,restart=8) # NEB8から計算を開始する
AutoVib
from grrmpy.automate import AutoVib
vib = AutoVib(ts_list)
vib.run()
vib.summary() # Summary.htmlに振動数の表を出力
vib.write_mode() # vibフォルダ内に虚数振動のtrajファイルが作成される
- class grrmpy.automate.auto_vib.AutoVib(ts_list, indices=None, calc_func=<function pfp_calculator>, name='vib', **kwargs)[ソース]
-
- write_mode(i=None)[ソース]
虚振動モードのtrajファイルを作成する
Parameters:
- i: int
- TS{i}の虚振動の振動モードでtrajファイルを作成するNoneを指定した場合,全てのTSの虚振動のtrajが作成される虚振動が確認できなかったTSのtrajファイルは作成されない
- forlder:str
- 指定したフォルダ内にtrajファイルが作成される
注釈
事前にsummary()を実行しておく必要がある.
>>> vib.summary() >>> vib.write_mode()
注釈
虚振動以外のモードを出力したい時,例えばTS5のmode=8の振動のtrajを作成する場合は
>>> vib[5].write_mode(8)
- class grrmpy.automate.queue.Queue(executor, priority=0, maxjob=inf)[ソース]
executorにクラスを指定して使用する. executorのクラスはrun()メソッドを必ず有する必要がある. さらにSTOP.txtで処理を停止し,途中再開できるようにするにはtodict()とfromdictのクラスメソッドを有する必要がある.
Parameters:
- executor:
- 計算実行クラスオブジェクト
- priority: int
- queueはタプルのリストであるが,階層の情報をリストの何番目に追加するかを指定する.priority=0の場合,階層の浅さが全てに優先されるため,幅優先になるpriority>0の時,深さ優先になる.
- maxjob: int
- maxjob回の計算を実行したら自動で計算を停止する.